来源:中国循证儿科杂志 作者:弓孟春 王慧君 卢宇蓝 葛小玲 周文浩
1. 概述
药物基因组学(Pharmacogenomics/Pharmacogenetics, PGx)的概念已存在数十年,其主要目的是明确个体间药物反应差异的重要基因变异。测序技术、生物信息学技术和计算技术的突破使得PGx研究在近两年来取得了令人瞩目的成就 [1, 2]。新的表型组构建技术为PGx研究提供了丰度和广度远超过传统研究的表型数据,使得在各类疾病中大量与药物相关的基因位点变异的意义被逐步阐释,包括:2型糖尿病[3-5]、高血压[6]、慢性充血性心力衰竭[7]、单核细胞增多症[8]、多发性硬化[9]、心律失常[10, 11]、急性淋巴细胞白血病治疗过程中糖皮质激素相关的骨坏死[12]及别嘌呤醇诱发的严重皮肤毒副反应等[13, 14]。最新在测序技术、医疗信息化及精准医学研究领域取得突飞猛进的进展,为PGx的临床部署创造了良好的条件[15]。PGx是目前将基因组信息整合入临床实践,提供临床决策支持的首选领域[16],其优势在于:①避免大量的与基因信息使用相关的伦理学难题;②专注于药物剂量制定,而不涉及疾病风险评估;③目前存在大量可用信息,且已被反复论证;④患者从改善临床预后、缩短疗程、减少治疗费用、减少药物副反应等方面获益显著;⑤欧美已纳入医保,具有广泛的使用案例基础及应用经验;⑥成熟的医疗信息技术解决方案,尤其是与之相关的临床决策支持系统(Clinical Decision Support System, CDSS),可有效促进临床的整合应用。
然而,尽管基础研究取得了长足发展,将PGx数据应用于临床实践的过程却非常缓慢。发达国家在PGx临床部署的实践中遇到的挑战包括:①缺乏药物基因组数据,诊疗中对遗传信息的抵触;②对于费用和保险支付的顾虑等[17-19]。只有当PGx在电子病历系统中保存,并在医嘱开立的当时触发相应规则及向临床医生呈递后续决策信息时,才能实现其最大应用价值。
本文从国外PGx临床部署的实践经验入手,着眼于在中国的大型教学医院进行PGx临床部署的顶层设计思考。
2. 国外PGx临床部署的现状
PGx的临床部署意义重大,PGx的临床部署为患者带来重大获益,避免大量的药物副反应的发生[20, 21],在目前已经进行的成本效益分析中体现出显著的优势[22]。美国98.5%的白人和99.1%的黑人携带至少一个高危的基因型[23, 24]。Mayo Clinic 在2011至2013年进行的研究[25]发现,超过70%的心内科常用药物剂量会受到个体间遗传变异的显著影响。美国FDA批准的约1000种药物中,100种可以成为PGx检测的靶点(除了那些根据体细胞突变而制定的靶向药物)。30种最常见的高危药物的处方量约为7亿4千万次[26]。在St. Jude儿童医院,1年期间就诊患儿的48%接受了至少一种PGx高危药物的治疗,在Vanderbilt医学中心的成人患者中,该比值为54%[27]。
PharGKB是全球最重要的药物基因组学知识库,在其内容基础上制定的临床药物基因组实施协作组(CPIC)指南是PGx临床部署遵循的主要规范[28]。目前国际上重要的致力于将CPIC指南与临床电子病历进行整合的协作研究包括转化药物基因组计划(TPP)[29]及eMERGE-PGx[30]。在此领域具有全球领先经验的机构包括:St. Jude儿童研究医院、Vanderbilt大学医学院、Mayo诊所、佛罗里达大学Shands医院和西奈山医学中心等。
St. Jude儿童研究医院早期采用单基因检测,主要针对TPMT和CYP2D6,用于指导硫唑嘌呤和可待因的剂量制定。2011年5月开始将基于芯片的PGx数据(230个基因的1936种位点变异)纳入临床信息系统,使得这些数据能够为临床医生提供实时决策支持。St. Jude儿童医院的目标是部署所有的CPIC指南药物-基因对。尽管这个项目目前是以科研项目的形式在实施,未来的目标是贯穿医疗服务全过程的标准化服务。
Mayo Clinic在eMERGE的辅助下,提出了RIGHT规范(Right Drug, Right Dose, Right Time-Using Genomic Data to Individualize Treatment)[31],使用NGS、PGRNseq和CYP2D6芯片来获取85个药物代谢相关基因的基因型结果,并将结果整合至电子病历;实施并不断开发针对高危药物基因组学结果的实时CDSS[32]。纳入临床信息系统的药物-基因对包括:HLA-B*1502-卡马西平、HLA- B*5701-阿巴卡韦、TPMT-硫唑嘌呤、IFNL3-干扰素、CYP2D6-可待因/他莫昔芬/曲马多、CYP2C19-氯吡格雷及SLCO1B1-辛伐他汀。
VUMC从2010年9月开始实施PREDICT项目,目前已有超过10000例患者接受了预先执行的基于Panel的药物基因学检测[26],包括SLCO1B1-辛伐他汀、CYP2C9 and VKORC1-华法令、CYP3A5-他克莫司和TPMT-硫唑嘌呤。对于首先入组的9589例患者的数据分析证实,在91%的受试者中发现了至少一个可以干预的变异(黑人中该比值高达96%)。
对国际上部署PGx的领先机构的实施过程进行分析,可以得出其成功的关键共性要素:①预先执行的基因检测策略(Preemptive, not reactive);将基因型检测及将检测结果迁移至电子病历作为一项系统工程来实施;②机构内部提供强有力的基础设施支持来辅助PGx的临床部署;③每家机构都设置了监管委员会,作为药理学及治疗学委员会的下属机构,以明确哪些基因检测结果可以进入电子病历,哪些药物要作为CDSS的靶点;④每家机构都有对所使用的基因-物对进行证据分析的过程,有些是在指南的辅助下进行的;⑤大部分选择了多基因panel,而非单个基因的检测(Multiple, not single);⑥各家机构均形成了完善的即时性CDSS,而又根据各家的具体临床实践进行了调整;⑦与时俱进,不断地加入新的临床意义显著的基因-药物对;⑧预先将受试者/患者知情同意进行到位;⑨各家机构均建立了完善的医生继续教育方案。这些经验对于中国的PGx临床部署具有重要的指导作用[33]。
3. PGx临床部署的方法论体系
3.1 预先执行策略
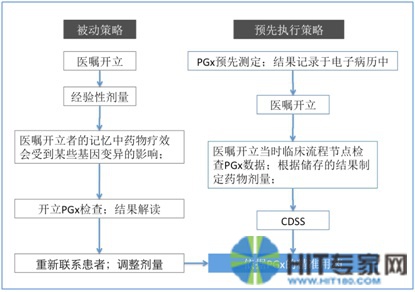
许多医疗机构采用“一个一个来”的策略对可能使用高危药物的患者进行特定基因检测,其检测结果后续可被用于与其他因素联合制定药物的剂量,如华法令的剂量制定策略中,同时使用遗传及非遗传因素来制定个体化的华法令用药剂量。然而,这种被动执行的策略有诸多弊端,如费用高昂、报告回复时间较长以至于可能延误医嘱开立、需要医生对于所使用药物与基因之间相互作用有足够的知识储备等。尽管此类单个基因的检测已在检验服务中存在已久,但临床应用一直推进缓慢。经常有高危的药物在没有进行药物基因组学评估之前被开立给那些可能出现治疗失败或严重不良反应风险极高的患者身上。
目前预先执行策略受到广泛的推崇,并逐渐成为临床实践规范[17, 34, 35]。是指在尚没有任何高危用药指证出现时,即进行多个与药物代谢相关的重要基因的检测,并将检测结果呈现在电子病历中,与其他临床信息一起构成处方开立时的决策参考因素。预先执行的多基因检测可以涵盖几乎所有的PGx重要基因,从而覆盖几乎所有的高危药物。检测的数据进入临床信息系统,在处方开立时,药物基因组信息与体重、身高、体表面积、性别等因素一起,为处方的决策提供指导,图1显示了被动策略与预先执行策略的实施中的主要差异[17, 36]。
PGx的临床部署为患者带来重大获益,避免大量的药物副反应的发生[20, 21]。在近期进行的一项针对大型教学医院的52942例患者的回顾性研究中[27]发现,由6类药物引起的副反应事件与药物代谢基因相关。如果使用预先执行的药物基因组筛查策略,可以避免至少398例严重不良反应事件,这为预先执行的多基因PGx在临床的广泛应用提供了强有力的证据[27]。将基因型信息整合入日常的临床药物决策制定过程可以改善患者安全,特别是当很多目标药物的相关高危基因变异在开立医嘱之前就已测出的话。
PGx的临床部署在目前已经进行的成本效益分析中体现出显著的优势[22]。预先执行策略的可行性已被多个医疗中心证实。来自VUMC PREDICT研究的结果显示:人群中>99%的个体至少携带一个可以干预的药物基因组变异;电子病历系统有能力将药物基因组变异信息整合入临床诊疗过程;相比于单个筛查的策略,预先执行策略可以显著减少检测次数[26]。
采用预先执行策略对医疗信息技术提出了重大挑战。预先得出的PGx数据需要保存在电子病历中,并在临床医生进行决策时提供实时支持。如果没有CDSS,基因检测的结果可能会被遗忘,而对患者造成重大风险,也为医疗机构的临床安全埋下巨大隐患,如在“已知”高危药物基因信息的情况下开立了高危药物,对患者造成严重损害。因此,在构建完善的相关CDSS之前,应避免采用预先执行及多基因同时进行的策略。
预先执行策略目前也在进一步完善中,以期精准定位高危人群,控制成本。Mayo诊所根据慢性疾病情况及人口学信息进行风险预测,从而精确地定位需要进行PGx检测的人群[37],使用去识别化的电子病历数据,构建了预测使用他汀、氯吡格雷及华法令的模型,模型纳入CDSS,用于向超过危险阈值的患者推荐预先执行的PGx检测。这是向精准定位目标人群的方向做出的有益尝试,也是对“预先执行”策略的重要补充。
3.2 组学数据向临床知识的转化
在临床常规工作中大规模实施PGx的前提是将组学数据翻译为已达成共识的临床操作规范,并构建经过审校的机器可读的PGx知识体系,形成电子病历中CDSS的构建规则。CPIC发布的指南是PGx信息化部署是目前最为重要的知识参考体系[38]。CPIC指南主要针对已达成广泛共识的药物-基因对,促进其临床部署。每个CPIC指南都遵循标准化的格式,并包含一个证据分级体系,用以明晰基因型-表性关联的可靠性及将该关联应用于医嘱开立时的证据强度。
PGx的临床部署需要医疗机构强有力的组织结构的支持。目前的成功经验之一是,组建PGx临床部署委员会对于适合本机构临床服务特点的基因-药物对进行分析,以决定是否将其迁移至电子病历系统。在审议的过程中,重要的参考依据是CPIC的指南。在将基因-药物对迁移至电子病历前,需要确认证据足够强,且具有实施相关临床决策支持的能力,特别是要评价电子病历的底层架构能否支持。对每一个基因型都要构建相应的翻译表,以将其解释为药物相关的表型信息,并预先确定这些表型的优先级、临床意义的解读报告、干预意见等。对于高危且明确可以干预的表型,应自动在电子病历中产生记录。
对于某一特定基因变异的临床可干预性评估体系目前是基于整合的独立于注释的消减算法(Combined Annotation-Dependent Depletion,CADD)计算的C评分[39]来构建的,克服了既往的注释和解读基因变异过程中存在的信息类型单一、广度不足等缺点。C评分与等位基因多样性、基因功能注释、致病性、疾病严重程度、实验验证的调控效应及复杂的性状关联等相关,C评分越高则表明变异的临床可干预性越强,是目前最先进的整合多维度评估基因变异的临床意义的体系,可在PGx的临床部署过程中提供临床可干预性评价。
3.3 临床决策支持系统
PGx的 CDSS实施可能遇到诸多挑战,包括数据储存和管理能力、系统集成水平及医生接受程度等。动态的干预性的临床决策支持通常有两种形式:验前警告和验后警告。当医生要开立特定的与某个药物-基因对相关的药物但并未进行基因检测时,系统触发警告,提示医生应该先完成基因检测(验前警告)。在大规模实施预先执行的PGx检测之前,需要使用这种验前警告系统来提示医生执行特定的药物基因检测。大规模实施预先执行的PGx检测后,提示医生该患者存在与当前开立的药物相关的基因信息(验后警告)。该警告会描述药物对患者的风险、提供替代用药或剂量调整的方案、要求修改或取消医嘱等[40]。
eMERGE-PGx协作组要求其9个参与中心实施PGx相关的CDS(临床决策支持),在每个机构都成功实施了至少一条PGx 的CDS。大部分采用了组学附属系统(OAS)来管理基因和基因组数据。所有的机构都将其电子病历内的CDS工具根据PGx知识进行了改进。对于实施过程中出现的某些延误案例进行的分析显示:造成部署延误的最常见原因不是PGx相关的问题。更为常见的是IT实施的问题,主要包括: 团队协作、沟通及人员配置,一般会导致系统上线时间总体推后约2个月,PGx的CDS项目并不比其他IT项目的实施难度大且大多是可以克服的[41]。目前已经有医疗机构尝试部署支持移动设备的基于PGx的CDSS[42],可以为临床医生和患者提供更大的便利。将基因组信息整合入临床诊疗过程及电子病历中可以极大的促进个体化医学的发展,实现的主要方式是基因信息驱动的临床决策支持。包括电子病历厂商、测序公司、患者权益组织等多个利益方参与是系统成功实施的关键[43]。
4. 实现PGx临床应用的基础设施
4.1 电子病历与基因组数据深度整合的跨机构信息共享平台
PGx近期的迅速发展,主要归功于计算技术辅助的表型化、基于多个异质性数据源的生物标记物发掘、评估临床可干预性的框架体系及具有高度战略互操作性的电子病历协作网络。eMERGE是一个多中心的医疗系统协作网络,其特点是涵盖了多个与电子病历数据整合的生物样本库[44]。eMERGE的主要研究目标是使用电子病历数据来发现新的遗传因素与疾病的相关性,同时探索将基因型数据和测序数据整合入临床实践的方法。eMERGE为协作中心提供以下的支持:①从不同电子病历中定义和提呈临床表型数据的专业支持;②将复杂的基因组数据跨中心的整合入统一的数据库;③将海量的基因组信息整合入电子病历并将这些数据与相关联的CDSS进行匹配;④就在临床诊疗中应用遗传学信息的内容对患者和医生进行教育。
通过部署一个针对84个药物基因的二代测序平台(RGRNseq系统),eMERGE-PGx计划[30]在1~3年的时间内评估9000例患者的PGx信息,将研究充分且证据充足的PGx表型数据纳入电子病历,用于构建临床决策支持体系,并评估实施的过程及临床效果。此外,将意义未明的药物基因组变异的数据存储于统一的数据中心,将基于电子病历收集的临床表型数据纳入,借助机器学习的技术,持续的发现新的PGx结果。eMERGE-PGx将构建一个公开的84个药物基因的测序数据库,称为SPHINX (http://emergesphinx.org),用以保存去识别化的基因组变异以及从电子病历中采集人口学信息、ICD代码、手术操作代码等。截止2015年2月eMERGE-PGx共分析了5000例患者。95.12%的变异其CADD评分超过20。96.19%的样本具有一条或多条CPIC指南的A级可干预指证。这些数据提示与药物代谢相关基因的分布广度,指出了在更广的范围内实施基于测序数据的用药的挑战,证实了实施精准医学的重要性[45]。
Li Li等[46]近期基于eMERGE的一项研究显示,使用临床表型特征的拓补学分析(患者-患者相似度)临床表现极为复杂、异质性极强的2型糖尿病临床亚型划分,为常见疾病基于电子病历等真实世界数据的临床亚型划分方法构建了独树一帜的方法论。而基于与eMERGE系统相关联的大量基因组变异信息,研究者有能力从不同的表型出发,探索相关的单核苷酸多态性(SNP)位点及相关的基因,并从信号转导通路等基础生物学研究角度来解释不同亚型之间的具有显著差别的临床表现,从而为这一常见疾病的亚型分组后的个体化治疗[47]及针对某一亚型的新药的研发[48]提供了新的途径。
4.2 测序技术的逐步完善及质量控制
基于Panel的测序技术是既往PGx数据的主要来源。全外显子组测序(WES)目前被广泛应用于人类遗传性疾病的分析,但其作为药物基因组分型的应用目前尚在探索阶段。对于下一代测序技术在PGx中的应用,有研究对于WES与基于Panel的技术之间进行了比较,证实了WES与MiSeq之间的一致性极高,证实WES是进行药物基因组分型的理想技术[49]。 中国人群中使用WES数据分析PGx与阿司匹林的疗效之间的关系研究也令人瞩目[50],该研究结果证实与阿司匹林的疗效及不良反应等相关的药物等位基因在中国人群中的分布具有明显的地域特点。基于SNP的 imputation也可能成为可行的PGx评估方法[51]。
4.3 机器可读的基因型结果及临床意义释读
基于PGx构建CDS是一项系统性工程,对于一份详尽的PGx报告,其中的信息需要被处理成机器可读的信息,并在电子病历中呈现[52]。信息部门基于这些信息构建CDS规则,可以借用常见的“合理用药”CDS模版(如青霉素与青霉素皮试结果之间的决策支持规则)。规则的内容由基因组变异解读委员会与来自多个学科的临床医生共同创建,并反复迭代。构建好的原型经过临床医生的审核,得到的反馈在后续的报警体系中予以体现。
需要建立持续的评估反馈机制来评价PGx的CDS警报在真实的医疗环境中的工作情况。使用自动化的数据采集及记录,可以分析警报触发时间、科室以及临床工作者的响应情况。每个触发并处理了警报的医生都被要求填写一份完整的问卷调查,以收集警报设计和内容方面的意见。
CPIC内已经评价、验证及审核的基因-药物对借助CDS在电子病历中进行实施[53]。将PGx成功在临床实践中部署,需要对PGx知识库进行审校,并翻译为机器可读的数据库,从而能够与具备CDSS的电子病历进行对接整合。目前电子病历厂商并没有将PGx作为常规的临床决策支持功能,因此CPIC的信息化工作组设计了一整套策略,以实现电子病历中PGx知识的应用。
需要对基因结果的呈现赋予更多的颗粒度。例如,CYP2C19*2与CYP2C19*17是同一基因的两个不同的变异,对氯吡格雷的药代动力学影响具有显著不同(前者为慢代谢,后者为快代谢)。但系统中只能表明是“CYP2C19基因异常”,无法呈现更多信息。需要医生点击进入报告查看界面才能看到更多信息。虽然增加颗粒度可以有利于设计更丰富的警报,但也需要进行大量的工作已完成检验结果的术语体系。
4.4 知识库的构建
目前重要的PGx知识库包括:PharmGKB,dbGaP和CinVar。PharmGKB将知识内容及时生成CPIC指南,并在指南与信息化整合的过程中给予了相应的指导意见,是目前最为重要的知识库。ClinVar则针对更大范围的基因变异纳入临床医生、病理科、药剂科等多个专业学科的临床解读,对基因变异与疾病的相关性、致病能力和可干预性进行注解。此外,另一个重要的信息来源是FDA的药品信息警告:8类药物目前明确在说明书中写入PGx检测的相关信息,包括三氧化二砷、Rasburicase、丙戊酸、阿巴卡韦、氯吡格雷、来那度胺、卡马西平、可待因。
目前尚缺乏针对中国人群数据的PGx知识库。可行的思路是梳理目前文献中已发表的以中国人群或东亚人群为主要对象的PGx研究,将其研究结果转化为临床干预方案。
4.5 表型组研究
在医疗系统完成跨机构信息互联互通及战略互操作的大背景下,PGx的进一步发展必须借助信息技术[54]。随着数据的广泛共享,医疗信息系统可以创造强大的知识生成环境,不断探索基因型-表型之间的关联信息。数据平台的应用使得基因变异的对于药物代谢、药物疗效和毒副作用等的影响可以嵌入到临床工作流程中。这样的系统即可基于基因型-表型PGx数据提供用药指导,也可以支持研究者发现更多的药物代谢相关的基因。
表型组与基因组的对应关系研究是近期的热点[55]。遗传学研究需要精准的表型定义,但电子病历的数据往往存储格式多样,记录完整性不一。表型组产生的过程中,Phenotyping所使用的NLP算法是至关重要的技术[56, 57]。eMERGE创立了13种基于电子病历的表型算法,在5个医疗中心(Group Health, Marshfield Clinic, Mayo Clinic, Northwestern University, and Vanderbilt University)的结构各异的电子病历中,成功开发、验证和部署了13种表型的NLP算法。在此基础上,eMERGE构建了表型化算法的标准及知识库PheKB[58],将成为PGx未来研究体系的重要支撑。
5. 实施PGx临床部署,临床医生的认识与态度
临床医生对于预先执行的药物基因组检测的态度,关乎PGx大规模临床部署项目的成败[59]。PREDICT研究组在一个教学医院中,评价了临床医生对于大范围PGx检测的实施的态度。受试者被问及的问题包括:影响基因检测及结果应用的主要因素、预期的效果以及将结果应用于临床服务的责任划分等。结果,所有临床医生均认同PGx对患者的药物治疗会产生影响。在出现明确基因-药物关联时,92%的医生希望得到即时的主动的通知。但对于由谁执行这个通知相关的剂量调整和是否应该通知患者这两个问题上,医生中存在分歧。这涉及临床责任的划分问题。
临床医生对于PGx知识自动化干预临床的态度也是各不相同[60]。在Mayo Clinic进行的一项研究[61]中,自2013年10月至今,研究组通过电子邮件,对医生在其临床实践中部署和使用PGx监测的看法进行了调查。研究主要针对的是医生对于PGx的总体看法,和他们是否认为PGx-CDS有用。CDS分为提醒医生注意的警报和提醒医生改变处方的警报。最后,使用规则的监测数据评估医生是否在警报的提醒下修改了医嘱。研究结果显示:近60位接受调查的医生中, 52%的医生并没有期待或并未明确他们是否会在未来的临床实践中使用PGx的信息。此外,53%的医生觉得警告信息“让人迷惑”、“惹人讨厌”或“让人无所适从”,或者难于找到进一步的信息。仅有30%的医生在接收到警告后改变了医嘱,换用了其他药物。该研究的结果显示临床医生对于PGx信息整合入临床实践感到不适。需要进一步优化PGx的CDS警告,以确保其可用性及用户友好度,从而让医生更积极地接受这些新的工具。
对于PGx相关内容的医生继续教育至关总要[62, 63]。医生对于PGx知识的欠缺是制约其临床应用的主要原因之一,这对于“被动策略”和预先执行策略都是类似的[64]。医学院教育体系的相关课程设置对于PGx的广泛应用也是至关重要的[65]。目前的医学教育体系中,关于PGx的临床应用的内容重视不足,导致对药物疗效产生重大影响的基因变异(如CYP2C19-氯吡格雷)重视不足。特定的专科中PGx知识的普及对于PGx的临床部署具有重要的推动作用,特别是各专科学术组织的推动作用[62]。
6. 构建中国PGx临床部署的顶层设计
全球范围内,在临床医学中创新性地使用基因组学正成为重要的发展方向[66],药物基因组学是目前条件最成熟的领域。随着国家对精准医学的科研及产业方面支持力度的加大,PGx临床部署的生态系统和基础设施将逐步得到完善。目前亟需在一个或数个机构内完成PGx临床部署的试点,以探索在伦理法规、测序技术、机器可读的PGx信息翻译、CDS构建、电子病历改造等过程中的技术难点及方法论,并将成本效益分析、医生及患者教育、基础技术研发等纳入研究体系,为多中心的预先执行的多基因PGx的大规模临床部署建立可推广的模型,引导精准医学临床实践的发展方向,积累中国人群的PGx数据及药物相关表型数据,建立表型-基因型深度整合的数据共享及分析平台,构建基于中国人群数据的药物基因组学知识库体系。
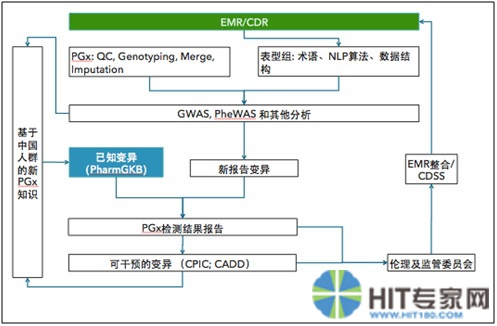
鉴于中国PGx临床部署尚处于起步阶段,借鉴国外的经验进行顶层设计十分必要,图2是在复习PGx临床部署文献基础上,结合中国PGx现状,构建总体框架,谨此作为本综述的总结和对中国PGx临床部署顶层设计思考。
(注:EMR 电子病历,CDR临床数据中心,PGx 药物基因组学,QC 质量控制,NLP 自然语言处理,GWAS 全基因组相关性分析,PheWAS 全表型组相关性分析,CDSS 临床决策支持系统,CPIC 临床药物基因组实施协作组,CADD 整合的独立于注释的消减算法)
【参考文献】
- Gross, E.R., et al., A personalized medicine approach for Asian Americans with the aldehyde dehydrogenase 2*2 variant. Annu Rev Pharmacol Toxicol, 2015. 55: 107-27.
- Hou, L., et al., Genetic variants associated with response to lithium treatment in bipolar disorder: a genome-wide association study. Lancet, 2016. 387(10023): 1085-93.
- Kho, A.N., et al., Use of diverse electronic medical record systems to identify genetic risk for type 2 diabetes within a genome-wide association study. J Am Med Inform Assoc, 2012. 19(2): 212-8.
- Zhou, K., et al., Pharmacogenomics in diabetes mellitus: insights into drug action and drug discovery. Nat Rev Endocrinol, 2016. 12(6): 337-46.
- Xu, H., et al., Validating drug repurposing signals using electronic health records: a case study of metformin associated with reduced cancer mortality. J Am Med Inform Assoc, 2015. 22(1): 179-91.
- Cooper-DeHoff, R.M. and J.A. Johnson, Hypertension pharmacogenomics: in search of personalized treatment approaches. Nat Rev Nephrol, 2016. 12(2): 110-22.
- Oeser, C., Pharmacogenetics: HCRTR2 gene is associated with response to HF therapy. Nat Rev Cardiol, 2016. 13(2): 64.
- Crosslin, D.R., et al., Genetic variation associated with circulating monocyte count in the eMERGE Network. Hum Mol Genet, 2013. 22(10): p. 2119-27.
- Davis, M.F. and J.L. Haines, The intelligent use and clinical benefits of electronic medical records in multiple sclerosis. Expert Rev Clin Immunol, 2015. 11(2): 205-11.
- Ritchie, M.D., et al., Genome- and phenome-wide analyses of cardiac conduction identifies markers of arrhythmia risk. Circulation, 2013. 127(13): 1377-85.
- Van Driest, S.L., et al., Association of Arrhythmia-Related Genetic Variants With Phenotypes Documented in Electronic Medical Records. Jama, 2016. 315(1): 47-57.
- Karol, S.E., et al., Genetics of glucocorticoid-associated osteonecrosis in children with acute lymphoblastic leukemia. Blood, 2015. 126(15): 1770-6.
- Li-Wan-Po, A., Pharmacogenetics begins to deliver on its promises. BMJ, 2015. 351: h5042.
- Ko, T.M., et al., Use of HLA-B*58:01 genotyping to prevent allopurinol induced severe cutaneous adverse reactions in Taiwan: national prospective cohort study. Bmj, 2015. 351: h4848.
- Gao, K. and J.R. Calabrese, Pharmacogenetics of lithium response: close to clinical practice? Lancet, 2016. 387(10023): 1034-6.
- Manolio, T.A., et al., Implementing genomic medicine in the clinic: the future is here. Genet Med, 2013. 15(4): 258-67.
- Dunnenberger, H.M., et al., Preemptive clinical pharmacogenetics implementation: current programs in five US medical centers. Annu Rev Pharmacol Toxicol, 2015. 55: 89-106.
- Roden, D.M., Cardiovascular pharmacogenomics: current status and future directions. J Hum Genet, 2016. 61(1): 79-85.
- Roden, D.M. and J.C. Denny, Integrating electronic health record genotype and phenotype datasets to transform patient care. Clin Pharmacol Ther, 2016. 99(3): 298-305.
- Saldivar, J.S., et al., Initial assessment of the benefits of implementing pharmacogenetics into the medical management of patients in a long-term care facility. Pharmgenomics Pers Med, 2016. 9: p. 1-6.
- Finkelstein, J., et al., Potential utility of precision medicine for older adults with polypharmacy: a case series study. Pharmgenomics Pers Med, 2016. 9: 31-45.
- Plumpton, C.O., et al., A Systematic Review of Economic Evaluations of Pharmacogenetic Testing for Prevention of Adverse Drug Reactions. Pharmacoeconomics, 2016.
- Pereira, N.L. and A.K. Stewart, Clinical Implementation of Cardiovascular Pharmacogenomics. Mayo Clin Proc, 2015. 90(6): 701-4.
- O’Donnell, P.H., K. Danahey, and M.J. Ratain, The Outlier in All of Us: Why Implementing Pharmacogenomics Could Matter for Everyone. Clin Pharmacol Ther, 2016. 99(4): 401-4.
- Kaufman, A.L., et al., Evidence for Clinical Implementation of Pharmacogenomics in Cardiac Drugs. Mayo Clin Proc, 2015. 90(6): 716-29.
- Van Driest, S.L., et al., Clinically actionable genotypes among 10,000 patients with preemptive pharmacogenomic testing. Clin Pharmacol Ther, 2014. 95(4): 423-31.
- Schildcrout, J.S., et al., Optimizing drug outcomes through pharmacogenetics: a case for preemptive genotyping. Clin Pharmacol Ther, 2012. 92(2): p. 235-42.
- Whirl-Carrillo, M., et al., Pharmacogenomics knowledge for personalized medicine. Clin Pharmacol Ther, 2012. 92(4): 414-7.
- Shuldiner, A.R., et al., The Pharmacogenomics Research Network Translational Pharmacogenetics Program: overcoming challenges of real-world implementation. Clin Pharmacol Ther, 2013. 94(2): 207-10.
- Rasmussen-Torvik, L.J., et al., Design and anticipated outcomes of the eMERGE-PGx project: a multicenter pilot for preemptive pharmacogenomics in electronic health record systems. Clin Pharmacol Ther, 2014. 96(4): 482-9.
- Bielinski, S.J., et al., Preemptive genotyping for personalized medicine: design of the right drug, right dose, right time-using genomic data to individualize treatment protocol. Mayo Clin Proc, 2014. 89(1): 25-33.
- Farrugia, G. and R.M. Weinshilboum, Challenges in implementing genomic medicine: the Mayo Clinic Center for Individualized Medicine. Clin Pharmacol Ther, 2013. 94(2): 204-6.
- Arwood, M.J., et al., Implementing Pharmacogenomics at Your Institution: Establishment and Overcoming Implementation Challenges. Clin Transl Sci, 2016.
- Carpenter, J.S., et al., Pharmacogenomically actionable medications in a safety net health care system. PeerJ, 2016. 4. DOI:2050312115624333.
- Jaja, C., et al., Preemptive Genotyping of CYP2C8 and CYP2C9 Allelic Variants Involved in NSAIDs Metabolism for Sickle Cell Disease Pain Management. Clin Transl Sci, 2015. 8(4): 272-80.
- Roden, D.M., et al., Pharmacogenomics: the genetics of variable drug responses. Circulation, 2011. 123(15): 1661-70.
- Schildcrout, J.S., et al., A prognostic model based on readily available clinical data enriched a pre-emptive pharmacogenetic testing program. J Clin Epidemiol, 2016. 72: 107-15.
- Caudle, K.E., et al., Incorporation of pharmacogenomics into routine clinical practice: the Clinical Pharmacogenetics Implementation Consortium (CPIC) guideline development process. Curr Drug Metab, 2014. 15(2): 209-17.
- Kircher, M., et al., A general framework for estimating the relative pathogenicity of human genetic variants. Nat Genet, 2014. 46(3): 310-5.
- Bell, G.C., et al., Development and use of active clinical decision support for preemptive pharmacogenomics. J Am Med Inform Assoc, 2014. 21(e1): e93-9.
- Herr, T.M., et al., Practical considerations in genomic decision support: The eMERGE experience. J Pathol Inform, 2015. 6: 50.
- Blagec, K., et al., Examining perceptions of the usefulness and usability of a mobile-based system for pharmacogenomics clinical decision support: a mixed methods study. 2016. 4: e1671.
- Hartzler, A., et al., Stakeholder engagement: a key component of integrating genomic information into electronic health records. Genet Med, 2013. 15(10): p. 792-801.
- Gottesman, O., et al., The Electronic Medical Records and Genomics (eMERGE) Network: past, present, and future. Genet Med, 2013. 15(10): 761-71.
- Bush, W.S., et al., Genetic Variation among 82 Pharmacogenes: the PGRN-Seq data from the eMERGE Network. Clin Pharmacol Ther, 2016.
- Li, L., et al., Identification of type 2 diabetes subgroups through topological analysis of patient similarity. Sci Transl Med, 2015. 7(311): 311ra174.
- Scheen, A.J., Towards a genotype-based approach for a patient-centered pharmacologic therapy of type 2 diabetes. Ann Transl Med, 2015. 3(Suppl 1): S36.
- Fu, S., et al., Phenotypic assays identify azoramide as a small-molecule modulator of the unfolded protein response with antidiabetic activity. Sci Transl Med, 2015. 7(292): 292ra98.
- Chua, E.W., et al., Cross-Comparison of Exome Analysis, Next-Generation Sequencing of Amplicons, and the iPLEX((R)) ADME PGx Panel for Pharmacogenomic Profiling. Front Pharmacol, 2016. 7: 1.
- Yang, L., et al., [Pharmacogenomics study of 620 whole-exome sequencing: focusing on aspirin application]. Zhonghua Er Ke Za Zhi, 2016. 54(5): 332-6.
- Almoguera, B., et al., Imputation of TPMT defective alleles for the identification of patients with high-risk phenotypes. Front Genet, 2014. 5: 96.
- Nishimura, A.A., et al., Development of clinical decision support alerts for pharmacogenomic incidental findings from exome sequencing. Genet Med, 2015.
- Hoffman, J.M., et al., Developing knowledge resources to support precision medicine: principles from the Clinical Pharmacogenetics Implementation Consortium (CPIC). J Am Med Inform Assoc, 2016.
- Potamias, G., et al., Deciphering next-generation pharmacogenomics: an information technology perspective. Open Biol, 2014. 4(7).
- Mosley, J.D., et al., Identifying genetically driven clinical phenotypes using linear mixed models. Nat Commun, 2016. 7: p. 11433.
- Newton, K.M., et al., Validation of electronic medical record-based phenotyping algorithms: results and lessons learned from the eMERGE network. J Am Med Inform Assoc, 2013. 20(e1): e147-54.
- Greene, D., et al., Phenotype Similarity Regression for Identifying the Genetic Determinants of Rare Diseases. Am J Hum Genet, 2016. 98(3): 490-9.
- Kirby, J.C., et al., PheKB: a catalog and workflow for creating electronic phenotype algorithms for transportability. J Am Med Inform Assoc, 2016.
- Peterson, J.F., et al., Attitudes of clinicians following large-scale pharmacogenomics implementation. Pharmacogenomics J, 2015.
- Laerum, H., et al., A taste of individualized medicine: physicians’ reactions to automated genetic interpretations. J Am Med Inform Assoc, 2014. 21(e1): e143-6.
- St Sauver, J.L., et al., Integrating pharmacogenomics into clinical practice: promise vs reality. Am J Med, 2016.
- Johansen Taber, K.A. and B.D. Dickinson, Pharmacogenomic knowledge gaps and educational resource needs among physicians in selected specialties. Pharmgenomics Pers Med, 2014. 7: 145-62.
- Romagnoli, K.M., et al., Bringing clinical pharmacogenomics information to pharmacists: A qualitative study of information needs and resource requirements. Int J Med Inform, 2016. 86: 54-61.
- Haga, S.B. and J. Moaddeb, Comparison of delivery strategies for pharmacogenetic testing services. Pharmacogenet Genomics, 2014. 24(3): 139-45.
- Perry, C.G., et al., Educational innovations in clinical pharmacogenomics. Clin Pharmacol Ther, 2016. 99(6): 582-4.
- Manolio, T.A., et al., Global implementation of genomic medicine: We are not alone. Sci Transl Med, 2015. 7(290): 290ps13.
 HIT专家网
HIT专家网



评论前必须登录!
注册